이것은 1학년 2학기때 시스템프로그래밍 기초라는 과목을 들으면서 구현한 것인데 개념과 함께 누군가에겐 도움이 되지 않을까 싶어 글을 쓴다.
나도 이 과제 덕분에 프로그래밍을 하는 데 있어 막힘도 많았지만 구현하였을 때의 쾌감을 다시 한번 느끼지 않았나 싶어 글을 남겨본다.
K-means Clustering 이란?
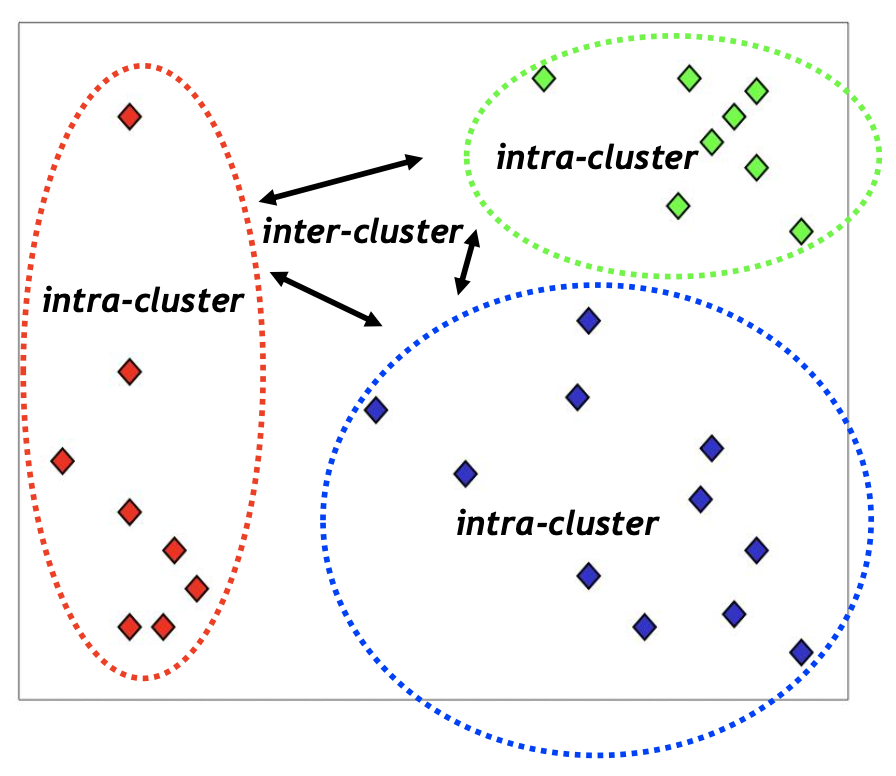
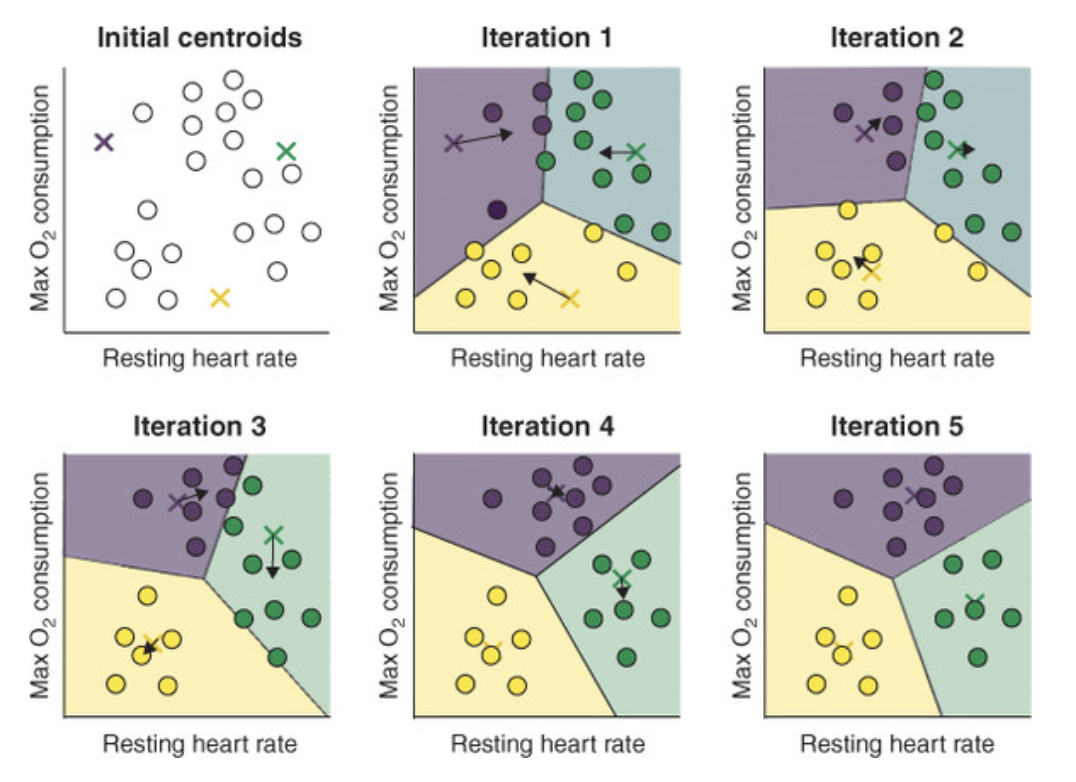
K-means Clustering은 비지도 학습(Unsupervised Learning)의 대표적인 알고리즘으로, 주어진 데이터를 K개의 Cluster(클러스터, 군집)으로 묶는 알고리즘이다.
K-means 알고리즘에서 K는 묶을 클러스터의 개수를 의미하고 means는 평균을 의미한다. 즉, 각 클러스터의 평균(mean)을 활용하여 K개의 군집으로 묶는다는 의미이다.
분류(Classification)와 군집(Clustering)의 차이
분류
분류는 지도학습(Supervised Learning) 방법에 속하며, 레이블(Lable)을 기반으로 데이터를 나누는 방법이다.
군집화
군집화는 비지도학습(Unsupervised Learning) 방법에 속하며, 레이블(Lable)이 주어지지 않은 상황에서 주어진 데이터를 묶는 방법을 의미한다.
문제 목표
Goal: organize n d-dimensional data into K clusters. (Each cluster has a center, which is called centroid)
즉, D-차원의 n개의 데이터가 주어지는데 이를 K개의 클러스터로 묶어야 한다.

나의 구현 방식
이 문제를 처음으로 곰곰히 생각해 봤을 때 드는 생각이.. 정답이 없지 않나? 였다. (당연하다..)
왜냐면 centroid(클러스터의 중심)를 어떻게 정하고 거리를 어떤 방식으로 정하는지에 따라 결과가 다르게 나오기 때문이다.
아니나 다를까 거리를 구하는 방식만 하더라도 이렇게 많다.. (참고)
그래서 교수님도 정답이 없긴 하지만 보고서와 발표를 참고하여 점수를 주신 것 같다.
결국 구현하는데 있어 정답은 없다. 나는 다음과 같은 방식을 사용했다.
ex) 2차원의 8개의 데이터를 3개의 클러스터로 묶기 (d = 2 / n = 8 / k = 3)
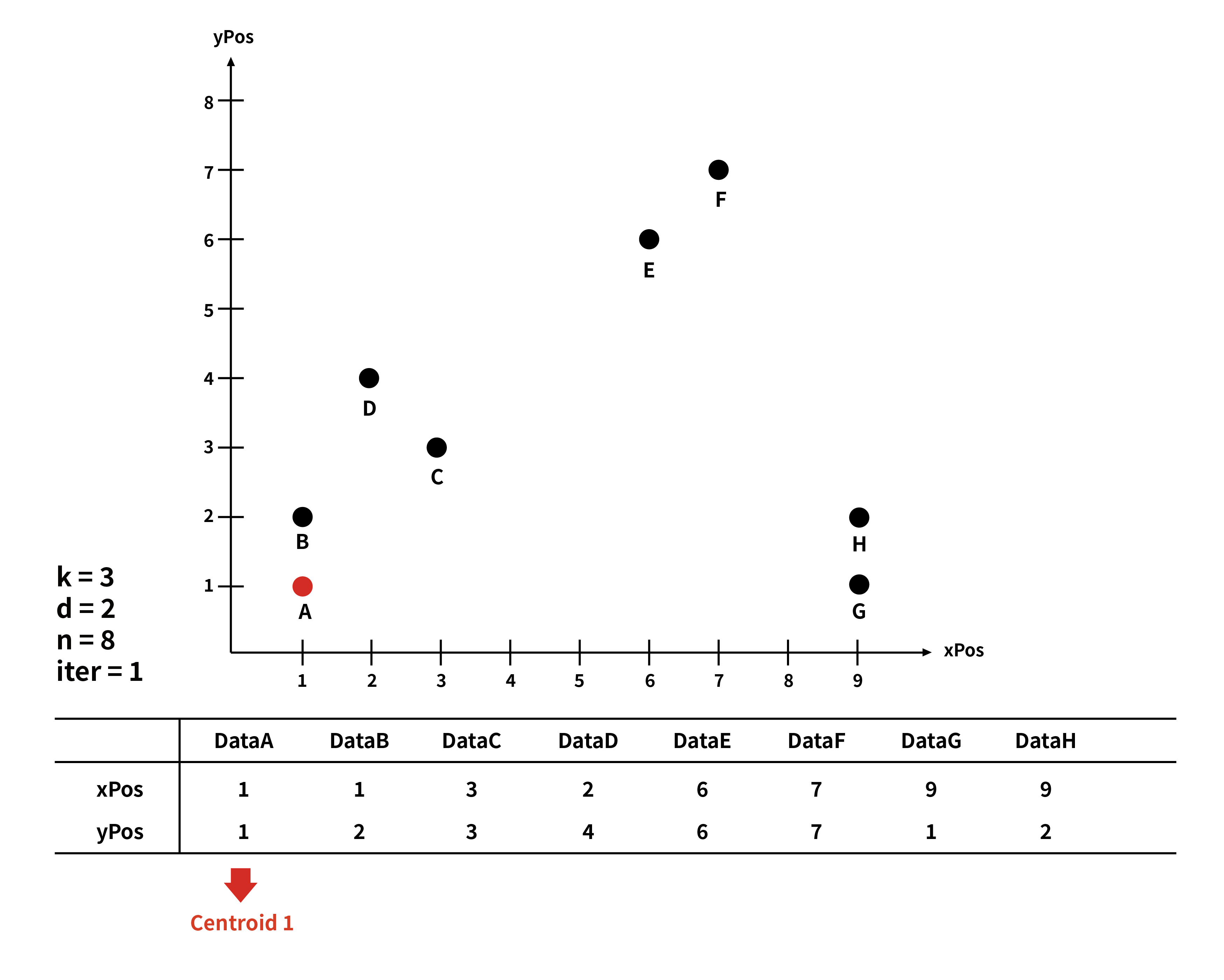
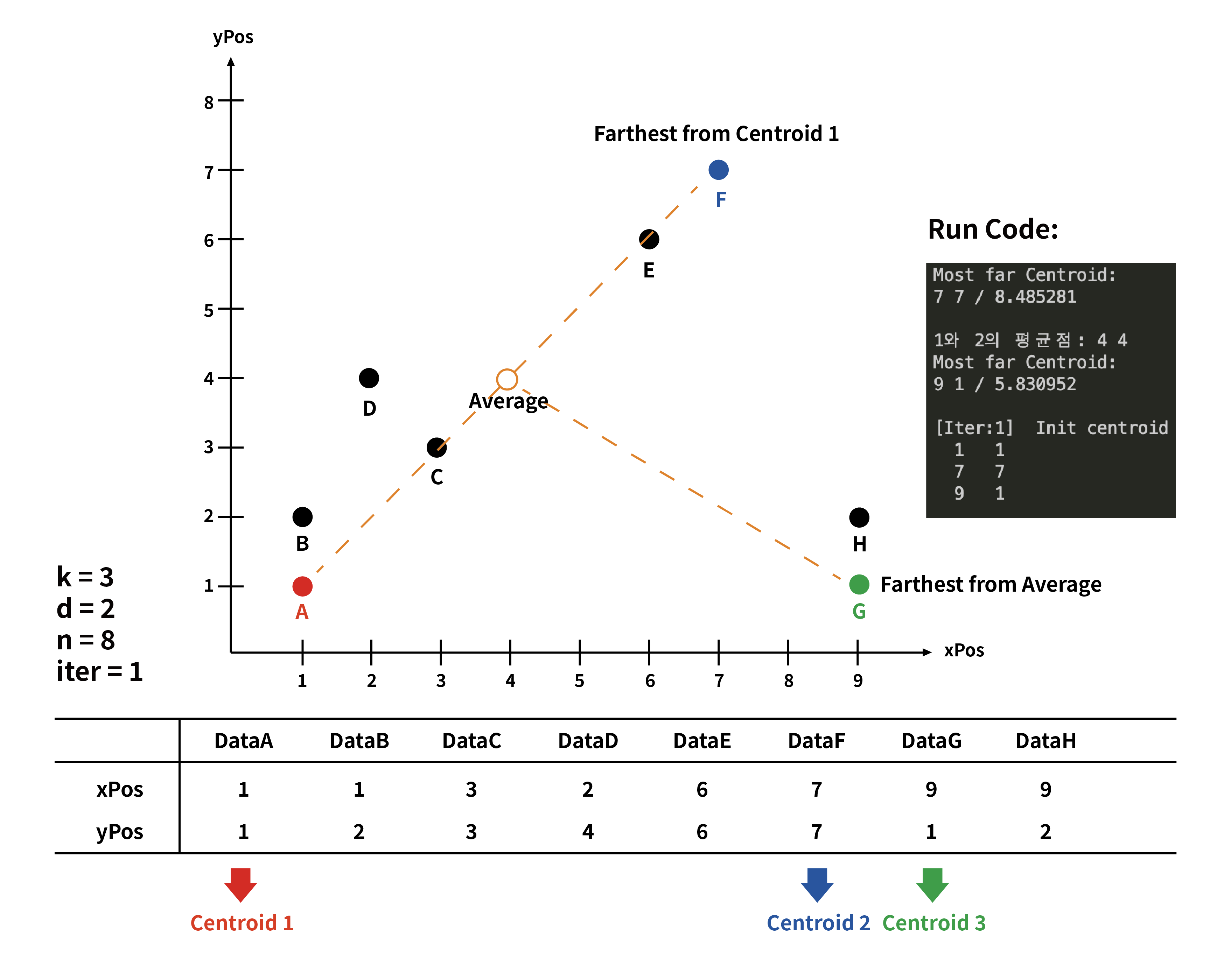
우선 centroid가 있어야 거리를 비교하여 클러스터를 생성할 것이니까, 1회차에서는 3개의 centroid를 정한다.(Init centroid)
1. 첫 centroid는 input 된 데이터 중 첫 번째로 들어온 값을 선정한다.
2. centroid가 된 A로 부터 가장 먼 거리에 있는 centroid를 다음 centroid로 정한다.
3. 두 centroid간의 평균점(Average)으로 부터 가장 먼 점이 마지막 centroid가 된다.
과정 3번은 조금 아쉬운 부분이 있다. 하지만 계산을 쉽게 하고 연산횟수를 줄이기 위해서 다음과 같은 방식을 사용하였다.
1. 평균점은 double 형이 아닌 int형으로 받아서 정확한 값이 아니라 상대적인 값이다.
2. 만약 4번째 centroid를 정한다면, 1,2,3 centroid의 평균점이 기준이 아니라, 1,2의 평균점과 3의 평균점으로 계산하였다.
k개의 클러스터를 묶을 centroid가 정해졌다면, 각각의 centroid 마다 모든 점의 거리를 측정해 준다.
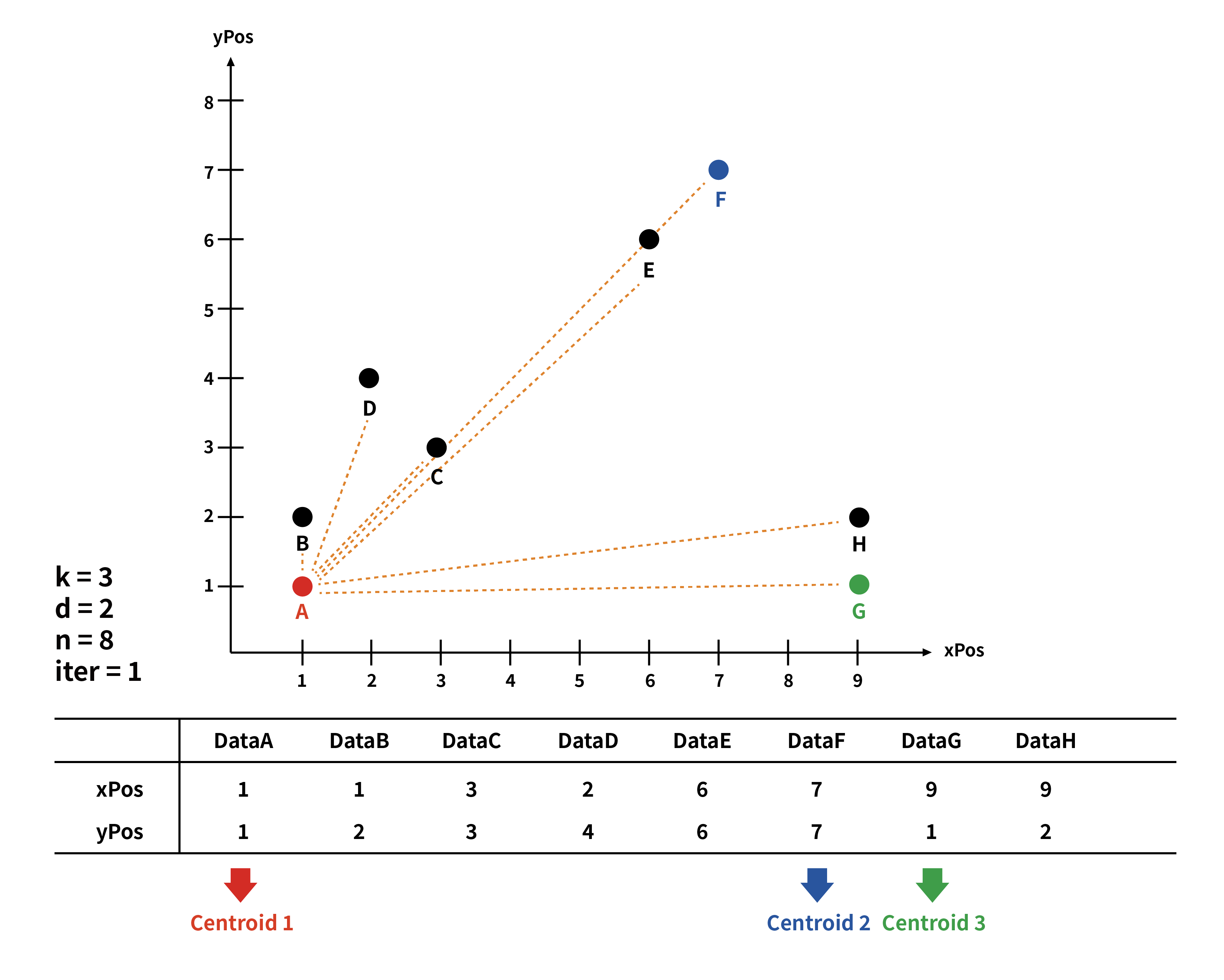
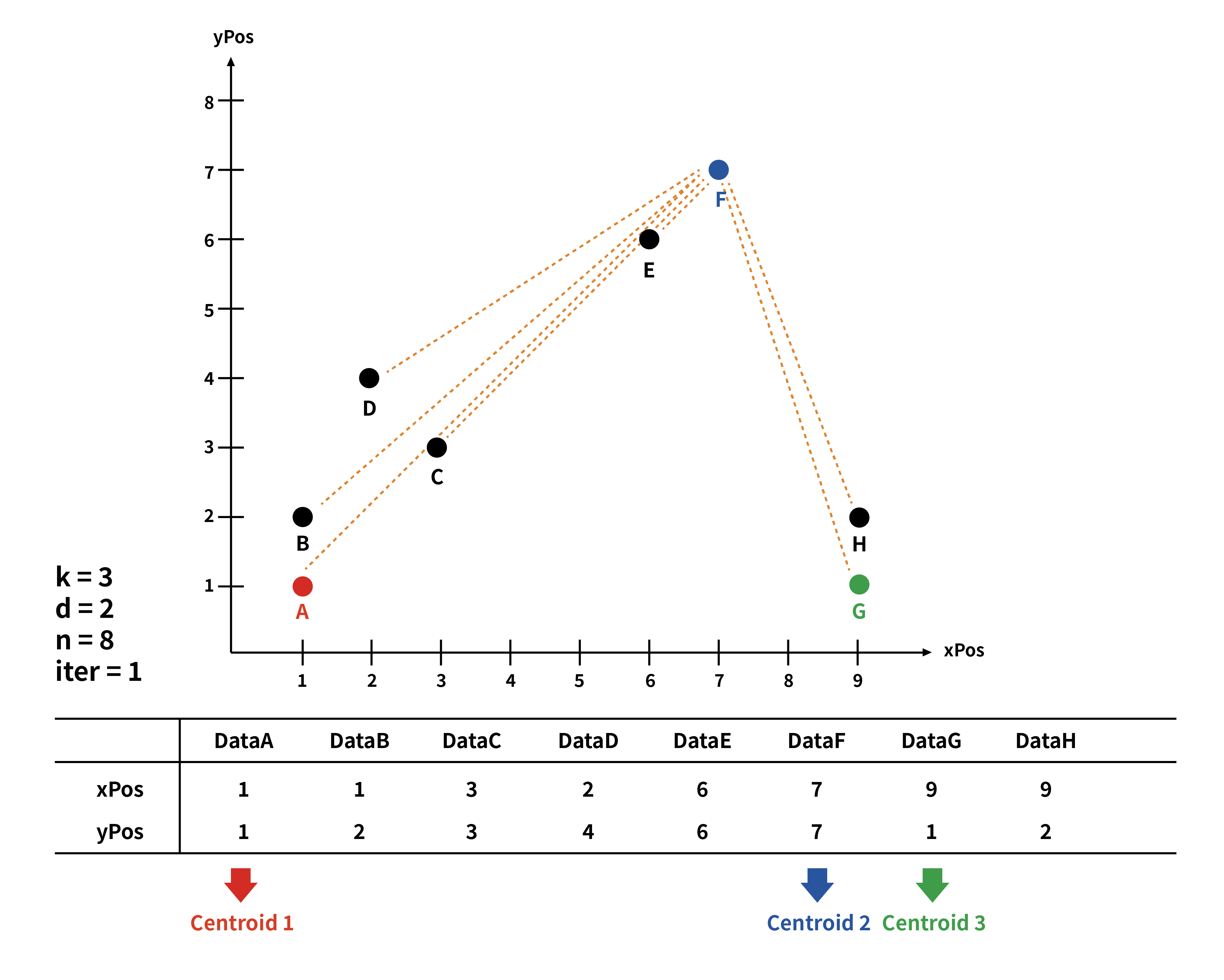
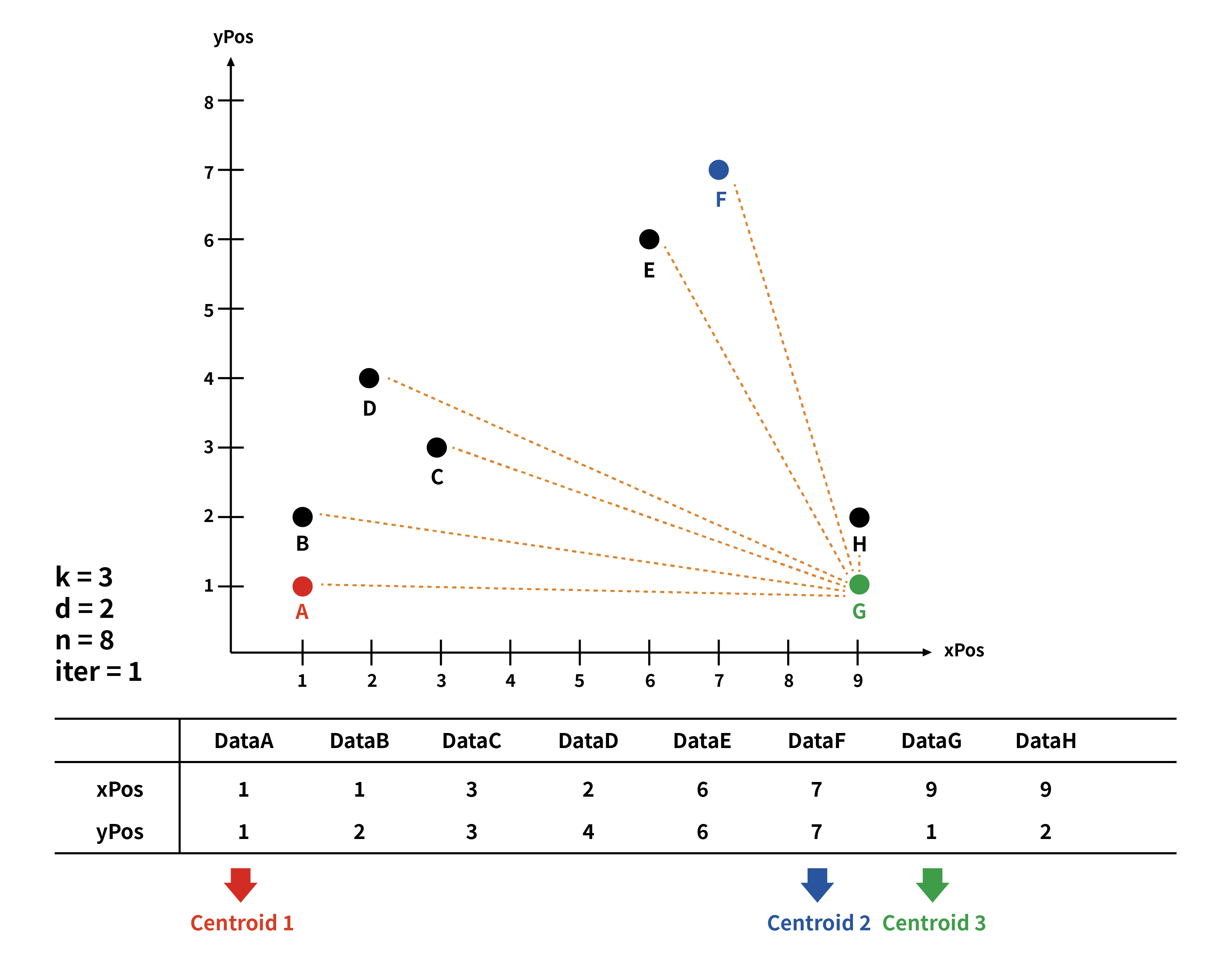
사진은 centroid를 기준으로 각각의 점들을 비교했다고 그렸는데, 실제로는 하나의 점마다 3개의 centroid를 비교하여서 가장 거리가 짧은 centroid를 구하였다.
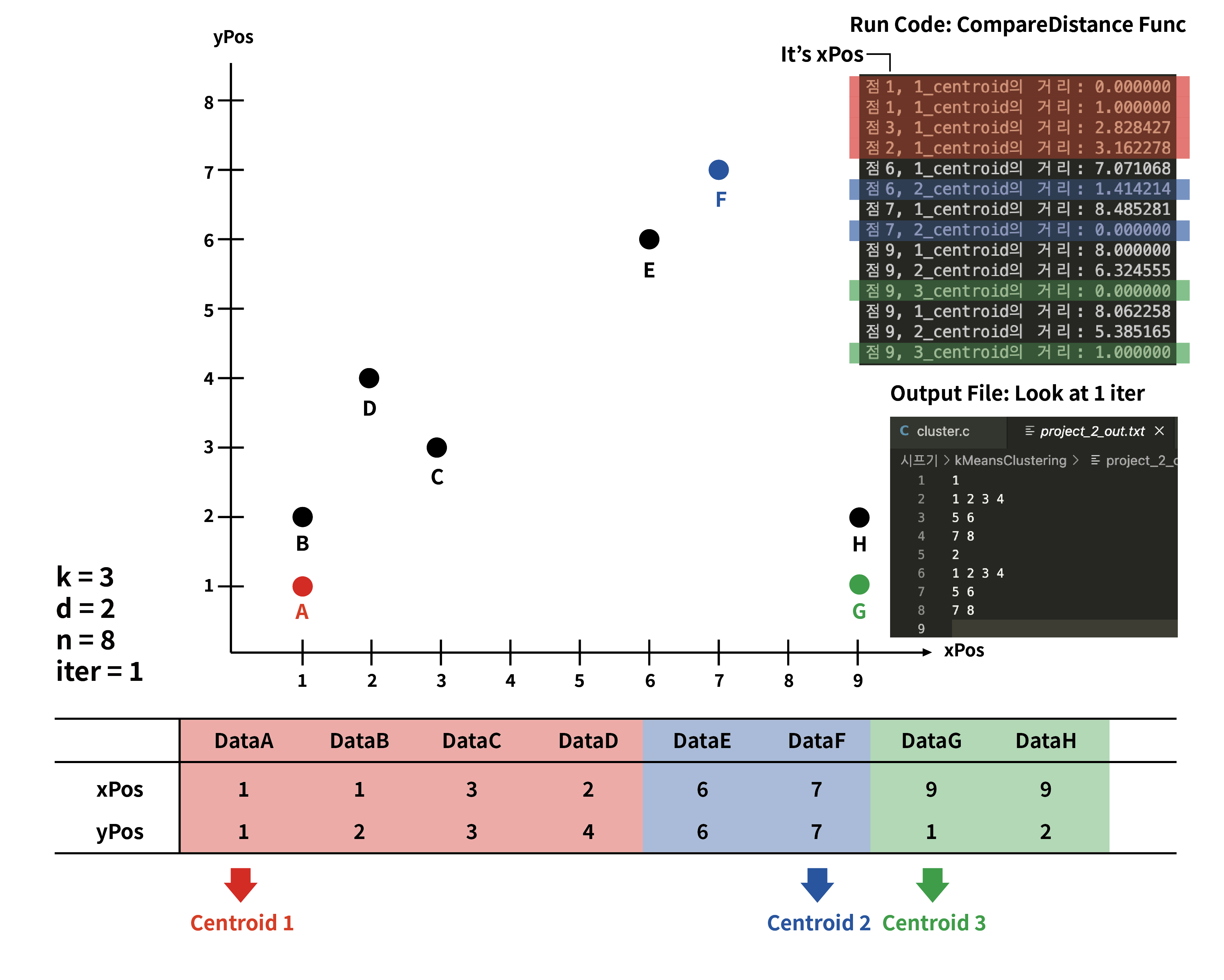

우측 결괏값을 print 한 것을 보면, 예를 들어 Data F점과 centroid 1,2,3을 비교하였는데 F와 centroid 2의 거리가 가장 가까워서 F점은 2 cluster에 포함된 것이다.
이런 식으로 분류하였을 때, project_2_out.txt 파일을 보면 각 클러스터에 점들을 들어온 순서로 묶어놨다. ,
1 # 1 회차
1 2 3 4 # cluster 1
5 6 # cluster 2
7 8 # cluster 3
# 다음과 같다.
1 # 1 회차
A B C D # cluster 1
E F # cluster 2
G H # cluster 3다음과 같이 클러스터를 나눌 수 있다.
이후 이제 3개의 클러스터가 탄생하였는데, 이 각각의 클러스터를 가지고 또 한 번 위와 같은 진행을 한다.
하지만 1회차와는 달리 2회차에서는 centroid를 정할 때 클러스터 별로 따로 centroid를 정한다.
즉 cluster 1의 점들은 자기들끼리 거리비교를 한 후 중심값과 가장 가까운 점을 클러스터로 삼는다.
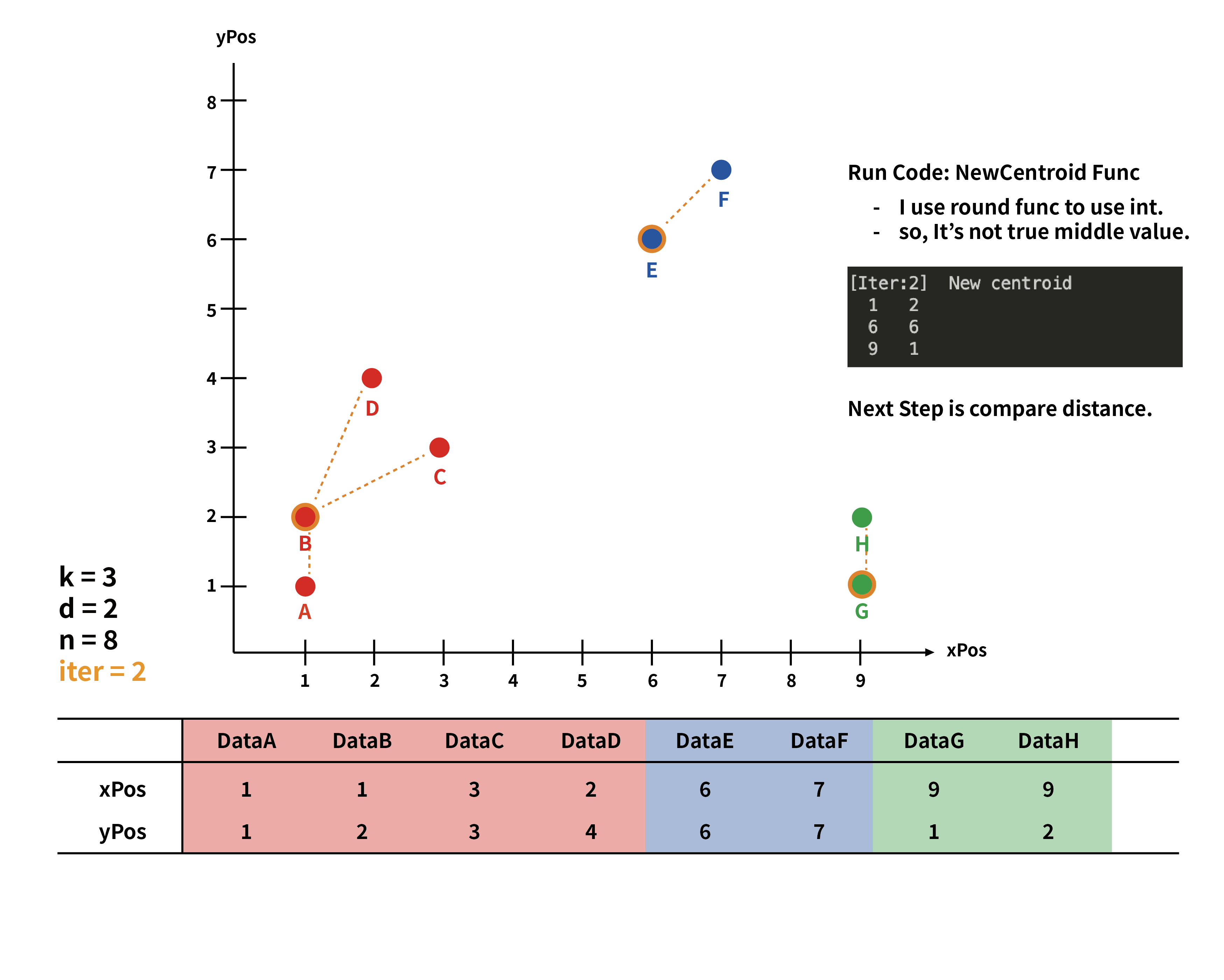
그랬을 때 2회차의 centroid는 B, E, G로 1회차 때와 다르게 나타났다.
하지만 2회차의 centroid로 클러스터를 만들어 내도 1회차때 project_2_out.txt 파일을 보면 결괏값이 같은 것을 볼 수 있다.
그렇기 때문에 더 이상 클러스트는 새로운 centroid를 구해도 동일한 결과와 동일한 클러스터 집합을 볼 것이기 때문에 종료한다.
느낀 점
해당 과제는 2번째 과제이고 1번째 과제를 할 때 matrix chain multiplication(mcm)을 구하는 것이었는데, 어떤 분이 내 코드를 보시곤 다음과 같이 하면 좋을 것 같다는 의견을 받았다.
- main 함수의 기능이 많다.
- 너무 단순한 변수명과 변수와 파라미터의 이름이 의미 없이 중복되면 코드를 분석하기 힘들어진다.
- 함수는 하나의 기능만 하도록 해라.
정말 여태까지의 코드를 짬에 있어서 엄청 인상 깊었던 피드백이었다.
이 이후로 나는 절차든 객체지향 방식이든 코드의 진행방식에 있어서 큰 발전이 있었다고 생각한다.
이 과제를 하면서도 다음과 같은 점들을 지키면서 하려고 노력했고 나름 만족할만하게 코드를 정리했다고 생각한다.
만약 내가 짠 코드를 읽기 힘들다고 한다면 위와 같은 점들을 지키면서 코딩해 보자.
내가 막혔을 때 다른 사람이 조언을 해주기도 훨씬 수월해진다.
다음 코드를 짜고 나서 조금 아쉬운 부분이 있었다면, 함수를 왔다 갔다 하다 보니.. 어떤 함수 내에서 동적할당한 변수가 다른 함수에서 쓰여서 프로그램이 종료될 때 동적해제를 못해주었다는 점이 아쉬웠다. 이 부분에 있어서 교수님께 질문을 하니 현재 내가 짠 구조에서는 수정하기 어렵다고 하셔서 나중에는 설계를 먼저 하고 짜는 것을 추천한다는 답변을 들었다..😅 들켰다
마지막으로.. 이 과제가 잘 안 풀려서 한 4일 동안 1시간씩 쪽잠 자면서 풀었던 기억이 난다.. 하지만 내가 쉽게 하지 못하는 것을 해내기 위해서 노력하였고 그 결과는 실력의 퀀텀점프 급이었다. 개발자로서 고민하고 더 좋은 코드를 만들려는 자세는 중요한 것 같다.
구현 코드
#include <stdio.h>
#include <stdlib.h>
#include <stdbool.h>
#include <math.h>
#define INPUT_FILE_NAME "project_2.txt"
#define OUTPUT_FILE_NAME "project_2_out.txt"
int iter = 1;
FILE *infp;
FILE *outfp;
typedef struct
{
int *pos;
} Data;
bool Finish(int k, int d, int n, int **centroid, int **prev_centriod)
{
// 이전 centroid 와의 같음을 판별함.
bool trig = false;
for (int i = 0; i < k; i++)
{
for (int j = 0; j < d; j++)
{
if (centroid[i][j] != prev_centriod[i][j])
trig = true;
}
}
return trig;
}
int **NewCentroid(int k, int d, int n, Data *data, int **cluster, int *cluster_len)
{
int **centroid = (int **)malloc(sizeof(int *) * k);
for (int i = 0; i < k; i++)
centroid[i] = (int *)malloc(sizeof(int) * d);
int sum;
int pos;
// 클러스터 갯수만큼
for (int i = 0; i < k; i++)
{
for (int j = 0; j < d; j++)
{
sum = 0;
pos = 0;
// 각각의 클러스터가 가지고 있는 점의 개수 만큼
for (int k = 0; k < cluster_len[i]; k++)
{
int idx = cluster[i][k] - 1;
sum += data[idx].pos[j];
}
pos = (int)round(sum / cluster_len[i]);
centroid[i][j] = pos;
}
}
// New centroid 출력
printf("[Iter:%d] New centroid \n", iter);
for (int i = 0; i < k; i++)
{
for (int j = 0; j < d; j++)
{
printf("%3d ", centroid[i][j]);
}
printf("\n");
}
printf("\n");
return centroid;
}
void PrintCluster(int k, int **cluster, int *cluster_len)
{
fprintf(outfp, "%d \n", iter);
for (int i = 0; i < k; i++)
{
for (int j = 0; j < cluster_len[i]; j++)
{
printf("%d ", cluster[i][j]);
fprintf(outfp, "%d ", cluster[i][j]);
}
printf("\n");
fprintf(outfp, "\n");
}
printf("\n");
}
int **CompareDistance(int k, int d, int n, Data *data, int **centroid, int *cluster_len)
{
int row = 0;
int sum = 0;
double min_distance = 99999999.0;
int min_centroid_idx = 0;
int **cluster = (int **)malloc(sizeof(int *) * k);
for (int i = 0; i < k; i++)
cluster[i] = (int *)malloc(sizeof(int) * 1);
int *cluster_cnt = (int *)calloc(k, sizeof(int));
// 정점 간 거리계산
for (int i = 0; i < n; i++)
{
for (int x = 0; x < k; x++)
{
sum = 0;
for (int y = 0; y < d; y++)
sum += pow(centroid[x][y] - data[row].pos[y], 2);
double distance = sqrt(sum);
if (distance < min_distance)
{
min_distance = distance;
min_centroid_idx = x;
// printf("점%d, %d_centroid의 거리: %lf \n", data[row].pos[0], (x + 1), distance);
}
}
cluster[min_centroid_idx] = (int *)realloc(cluster[min_centroid_idx], sizeof(int) * (cluster_cnt[min_centroid_idx] + 1));
cluster[min_centroid_idx][cluster_cnt[min_centroid_idx]] = ++row;
cluster_cnt[min_centroid_idx]++;
min_distance = 99999999.0;
}
PrintCluster(k, cluster, cluster_cnt);
iter++;
// 아쉬운 점: main으로 cluster_length를 보내기 위함.
// -> 이렇게 하지 않으면 main 에서 while을 통해 또 cluster의 길이를 카운팅 해야함.
for (int i = 0; i < k; i++)
cluster_len[i] = cluster_cnt[i];
return cluster;
}
int *MostFarCentroid(int k, int d, int n, Data *data, int *centroid[])
{
int row = 0;
int sum = 0;
double max_distance = 0;
int *max_distance_pos = (int *)malloc(sizeof(int) * d);
// 정점 간 거리계산
for (int i = 0; i < n; i++)
{
sum = 0;
for (int y = 0; y < d; y++)
sum += pow(centroid[0][y] - data[row].pos[y], 2);
// 거리 비교 후 centroid와 가장 먼 정점을 넣음
double distance = sqrt(sum);
if (max_distance < distance)
{
max_distance = distance;
for (int i = 0; i < d; i++)
{
max_distance_pos[i] = data[row].pos[i];
}
}
row++;
}
printf("Most far Centroid: \n");
for (int i = 0; i < d; i++)
printf("%d ", max_distance_pos[i]);
printf("/ %lf \n", max_distance);
printf("\n");
return max_distance_pos;
}
int **InitCentroid(int k, int d, int n, Data *data, int *cluster_len)
{
// 클러스터 x번째
int x = 0;
// 클러스터 개수만큼 centroid의 좌표를 담을 배열을 동적할당하여 만듬.
int **centroid = (int **)malloc(sizeof(int *) * k);
for (int i = 0; i < k; i++)
centroid[i] = (int *)malloc(sizeof(int) * d);
int **aver_centroid = (int **)malloc(sizeof(int *) * 1);
aver_centroid[0] = (int *)malloc(sizeof(int) * d);
// 반복 첫번째에는 centroid를 맨 처음 값으로 사용
centroid[0] = data[0].pos;
// 초기 centroid 저장
for (int i = 0; i < k - 1; i++)
{
if (x == 0)
{
centroid[i + 1] = MostFarCentroid(k, d, n, data, centroid);
for (int j = 0; j < d; j++)
aver_centroid[0][j] = centroid[i][j];
}
else
{
printf("%d와 %d의 평균점: ", i, i + 1);
for (int j = 0; j < d; j++)
{
aver_centroid[0][j] = (int)round((aver_centroid[0][j] + centroid[i][j]) / 2);
printf("%d ", aver_centroid[0][j]);
}
printf("\n");
centroid[i + 1] = MostFarCentroid(k, d, n, data, aver_centroid);
}
x++;
}
// Init centroid 출력
printf("[Iter:1] Init centroid \n");
for (int i = 0; i < k; i++)
{
for (int j = 0; j < d; j++)
{
printf("%3d ", centroid[i][j]);
}
printf("\n");
}
printf("\n");
return CompareDistance(k, d, n, data, centroid, cluster_len);
}
int main(void)
{
int k, d, n;
int ch;
int row = 0, col = 0;
int **centroid = (int **)malloc(sizeof(int *) * k);
int **prev_centroid = (int **)malloc(sizeof(int *) * k);
for (int i = 0; i < k; i++)
prev_centroid[i] = (int *)calloc(d, sizeof(int));
int **cluster = (int **)malloc(sizeof(int *) * k);
int *cluster_len = (int *)calloc(k, sizeof(int));
infp = fopen(INPUT_FILE_NAME, "r");
outfp = fopen(OUTPUT_FILE_NAME, "w");
if (infp == NULL)
{
printf("\n Unable to open file! \n");
return 0;
}
// k = 클러스터의 개수
// d = 차원, 좌표의 개수
// n = 데이터의 개수
fscanf(infp, "%d %d %d", &k, &d, &n);
// 데이터의 개수 만큼 구조체 배열을 만듬
Data *data = (Data *)malloc(sizeof(Data *) * n);
// 구조체 i번째에 좌표를 넣을 칸을 동적할당함.
for (int i = 0; i < n; i++)
data[i].pos = (int *)malloc(sizeof(int *) * d);
while (EOF != fscanf(infp, "%d ", &ch))
{
data[row].pos[col] = ch;
col += 1;
if (col == d)
{
col = 0;
row += 1;
}
}
cluster = InitCentroid(k, d, n, data, cluster_len);
while (true)
{
centroid = NewCentroid(k, d, n, data, cluster, cluster_len);
// 이전 centroid값과 현재 centroid 값이 같다면 종료
if (Finish(k, d, n, centroid, prev_centroid) == false)
{
printf("[FINISH] Same Centroid \n");
exit(0);
}
prev_centroid = centroid;
cluster = CompareDistance(k, d, n, data, centroid, cluster_len);
}
free(data);
free(cluster);
free(cluster_len);
free(centroid);
}'개발 > C' 카테고리의 다른 글
| 포인터 관련하여 정리 (0) | 2021.11.15 |
|---|---|
| C언어에서 선언된 배열 값외의 배열 주소에 접근이 가능할까? (0) | 2020.09.05 |